从NCBI上下载数据的办法
一、相关工具的下载
0 NCBI 简介
SRA(Sequence ReadArchive)数据库是用于存储二代测序的原始数据,包括 454,Illumina,SOLiD,IonTorrent,Helicos 和 CompleteGenomics。除了原始序列数据外,SRA现在也存在raw reads在参考基因的比对信息。
NCBI 上的 SRA 数据集可以分成四类:
- Studies:研究课题。研究课题可能包含多个Experiments。
- Experiments:实验设计。实验设计包含Sample、DNA source、测序平台、数据处理等信息,一个设计可能包含了多个runs。
- Samples:样品信息。
- Runs:测序结果集。测序仪器运行所产生的reads。
以上数据集自上至下具有包含关系
SRA 数据库用不同前缀区分数据,ERP 或 SRP 表示 Studies;SRS 表示 Samples;SRX 表示 Experiments;SRR 表示 Runs^0。
1 下载与安装
首先需要下载和安装两个官网上的工具 Entrez Direct 和 SPR toolkit,这两个软件在 Ubuntu 的商店里没办法搜到,下载安装方法是这样的:
1.1 Entrez Direct 的下载与安装
官网(https://www.ncbi.nlm.nih.gov/books/NBK179288/)上给出了几种安装方法,我尝试了第二种:

运行上述任意一条命令,可能弹出下述提示信息:

按提示输入命令,然后重新运行上述命令:
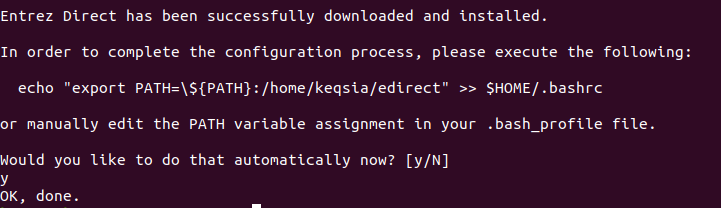
会提示你完成设置,按字面上大概意思可能是添加工具到环境变量,输入y即可。不过非常奇怪的是,按照网上的说法,这个工具包含了esearch命令,可以用esearch -help查看详情,但是我输入命令后却显示:
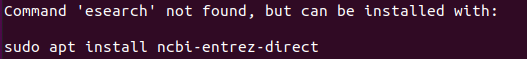
这个意思是之前没安装吗?关于这个问题,网上没找到答案:cry: ,所以我选择运行一下这个命令看看:
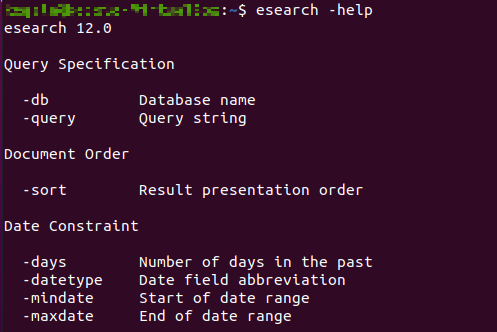
可以看到,安装后,帮助弹出终于成功了。下面是edirect工具的最常用命令:
| 工具名称 | 工具用途 | 常用参数 |
|---|---|---|
| esearch | 搜索命令,将所要检索的内容提交到 Entrez 中,返回相应的结果记录 | -db、-query |
| efetch | 下载 NCBI 数据库中的记录和报告并以相应格式打印输出 | -db、-id、-format、-mode |
| einfo | 获取目标结果在数据库中的信息 | -db、-dbs、-fields、-links |
| elink | 对目标结果在其他数据库中比配结果 | -db、-id、-related、-target、-name |
| epost | 上传 UIDs 或者 序列登记号 | -db、-id、-format、-input、-label |
| efilter | 对之前的检索结果进行过滤或限制 | -query、-sort、-field |
| xtract | 将esearch获得的 XML 格式结果转换成表格格式 | -pattern、-if、-block、-element、-sep、-filter |
| esummary | 获得 XML 格式的建立 | -db、-id、-format、-mode |
| ecitmatch | 统计引用数据 | -journal、-year、-volume、-page、-author |
1.2 SRA Toolkit 的下载与安装
SRA Toolkit 在官网上提供压缩包安装,与 Entrez Direct 工具安装方式不同,可以直接在虚拟机下载该包,也可以主机上下载后传输给虚拟机安装,这里采用vBOX提供的共享文件夹的方法传输安装包:
选择共享文件夹选项:
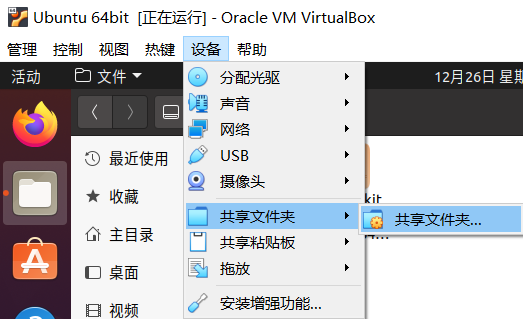
点击右侧添加共享文件夹,指定文件夹路径(选一个位置新建文件夹指定),勾选自动挂载(随启动直接加载)和固定分配,然后确定:
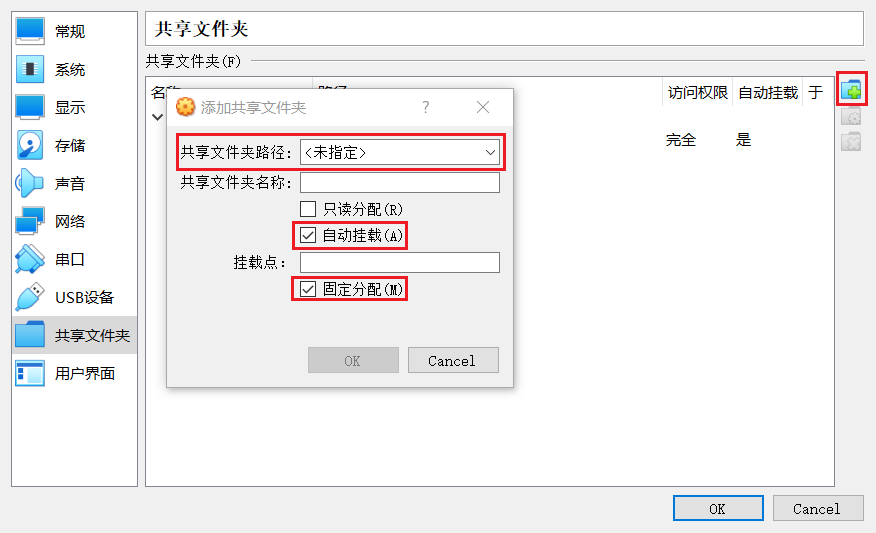
此时已经可以在文件管理器左栏看见共享文件夹了,但是点击会显示没有访问权限:
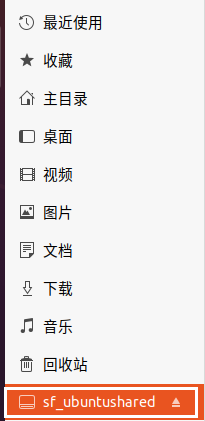
在终端中运行下面的命令,将目前的用户追加到文件的权限组中,然后重启系统^3:

将下载到的安装包复制到共享文件夹,现在共享文件夹中已经能看到安装包了:
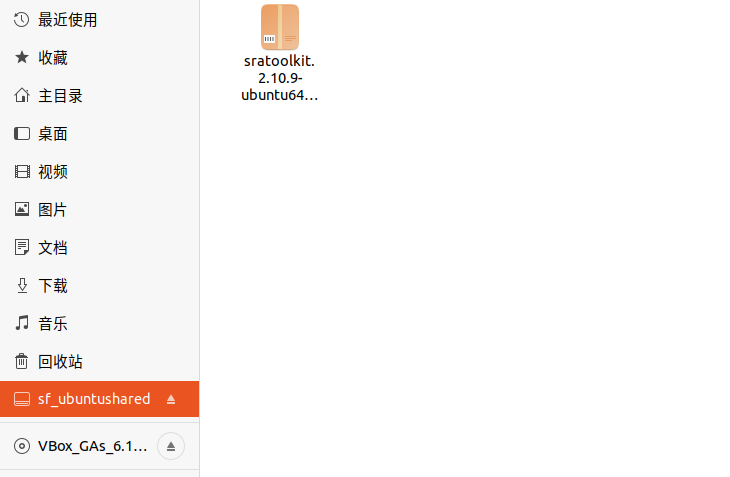
将文件复制到用户目录下,直接用
tar -zxvf sratoolkit.2.10.9-ubuntu64.tar.gz命令进行解压,可以看到,相应软件已经在用户目录下了:
下面将bin中的命令添加到环境变量(注意 Ubuntu 和自己安装的版本一致,调整代码):
1
echo "export PATH=\${PATH}:/home/keqsia/sratoolkit.2.10.9-ubuntu64/bin">>$HOME/.bashrc
注意每次修改.bashrc或设置环境变量后,使用
source ~/.bashrc使之立刻生效。.bashrc 文件在用户每次打开终端时都会执行一次,从而完成环境变量写入;有些教程会追加到 .profile 文件中,profile 在系统登录后执行,只在登录系统时执行一次,包括针对系统的/etc/profile和针对用户的 ~/.profile;bashrc 包括针对系统的 /etc/bash.bashrc 和针对用户的 ~/.bashrc。^2
有意思的是,运行
prefetch -h时产生了下面提示,要求你进行设置^3: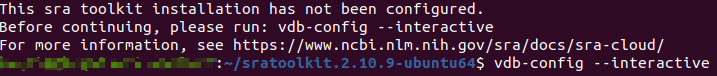
网上的解释是这样的^4:
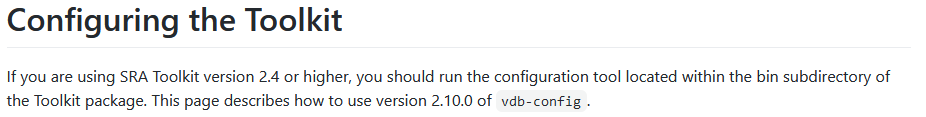
设置界面是这样的(博主未来有机会再探索):
设置完成后再运行prefetch -h,发现已经没有障碍了:
2 SRA Toolkit 的使用
下载数据列表:
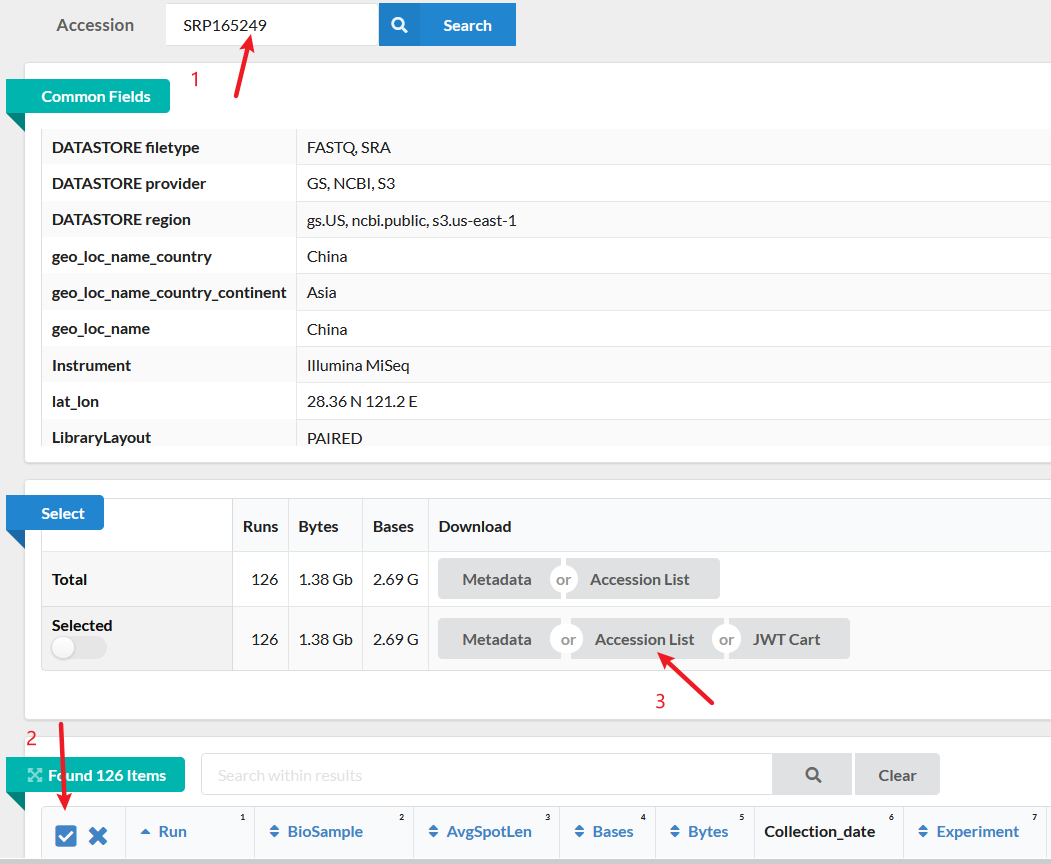
指定上一步下载的数据列表下载数据^5:
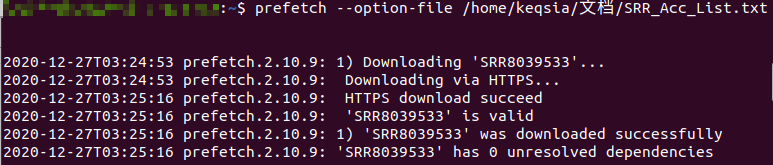
拉取完毕的数据以文件夹形式存放在用户目录,内部文件格式为sra。
参考文献:

